Actualidad informática
Noticias y novedades sobre informática
Por primera vez se simula en ordenador el genoma de un ser vivo
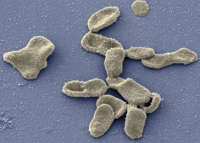
En una movida que promete traer las ventajas de diseño asistido por ordenador (CAD) a los ingenieros genéticos, el primer modelo de computadora de una bacteria completa se ha producido en los EE.UU.. Esto significa que los investigadores pronto será capaces de modificar los modelos de genoma de un organismo en una pantalla de ordenador – o crear formas de vida artificiales – sin los riesgos de llevar a cabo la biología en los laboratorios de biotecnología segura.
El agente patógeno se llama Mycoplasma genitalium, una bacteria implicada en una serie de infecciones uretrales y vaginales. Según los investigadores de la Universidad de Stanford en California, debido a que tiene el genoma más pequeño de cualquier organismo vivo, con sólo 525 genes. Por el contrario, el patógeno popular de laboratorio E. coli tiene 4288 genes.
La modelación se llevó a cabo por el bioingeniero Markus Covert y sus colegas. Para obtener los datos en bruto para su modelo, llevó a cabo una revisión exhaustiva de la literatura – que abarca 900 artículos de investigación – para permitir que el programa modelara 1900 comportamientos observados experimentalmente y las interacciones moleculares que M. genitalium puede tomar parte en durante su ciclo de vida.
En términos de software, se encontraron con los comportamientos de 525 genes podrían ser descritos por 28 algoritmos, cada uno rige el comportamiento de un módulo de software de modelado de un proceso biológico diferente. «Estos módulos se comunican entre sí después de cada paso de tiempo, para hacer un todo unificado que igualan en el mundo real el comportamiento de M. genitalium«, afirma el equipo de Stanford en un comunicado. Su investigación aparece en la revista Cell (doi: 10.1016/j.cell.2012.05.044).
Estos modelos en última instancia, darán a los biólogos la libertad de emprender «¿qué pasaría si» los escenarios más comunes en la ingeniería -el cambio de parámetros en el diseño del genoma, dicen, al igual que un ingeniero civil ajusta el ancho de tablero de un puente en una computadora para ver qué pasa. Además de ser experimentalmente útil, permitiendo organismos artificiales y formas de vida sintéticas que se creen virtualmente (sin daño a nadie), sino que también podría impulsar la seguridad de la biotecnología mediante la prevención de las creaciones accidentales de patógenos letales. En el año 2001, por ejemplo, investigadores en Australia accidentalmente crearon una cepa mortal de la viruela del ratón.
En un artículo de comentario en Cell, los sistemas de los biólogos Peter Freddolino y Saeed Tavazoie de la Universidad de Columbia dicen que esperan que el trabajo pronto se extenderá a los organismos de laboratorio de uso más común como la E. coli -, pero también advierten que la precisión de la técnica aún no se ha demostrado. No está claro, dicen, «qué tan bien los comportamientos generales se puede predecir a partir de una colección de parámetros obtenidos por separado», deducidas a partir de cientos de trabajos de investigación.
Pero el Institutos Nacionales de Salud de EE.UU., que financió el trabajo de modelado, está entusiasmado. Se cree que el modelo es un gran paso hacia la búsqueda de «nuevos enfoques para el diagnóstico y tratamiento de la enfermedad», dice James Anderson, director del programa.
Fuente: NewScientist
Codifican memoria regrabable en el ADN
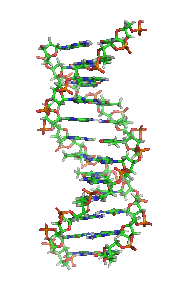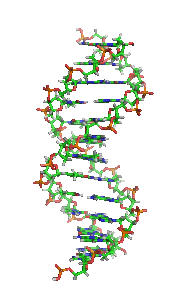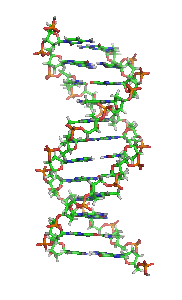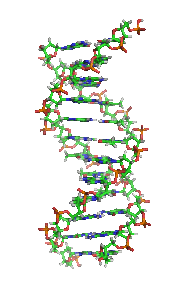
Investigadores han codificado una forma de memoria regrabable en el ADN. Intentaron crear un sistema de memoria regrabable al dividir elementos genéticos de un bacteriófago -un virus que infecta bacterias- en el ADN de la bacteria Escherichia coli. El artículo fue publicado en Procedings of the National Academy of Sciences y reseñado en Nature.
El uso de sistemas biológicos sintéticos en investigación, asistencia sanitaria, y fabricación, a menudo requiere de la comportamiento histórico autónomo dependiente y por lo tanto, alguna forma de ingeniería de memoria biológica. Por ejemplo, el estudio o la reprogramación de envejecimiento, el cáncer o el desarrollo se beneficiarían de los contadores codificados genéticamente capaces de grabar hasta la división celular de varios cientos o fenómenos de diferenciación. Aunque el material genético en sí mismo constituye un medio natural de almacenamiento de datos, herramientas que permitan a los investigadores de forma fiable y reversible escribir información en el ADN in vivo son escasas. En este sentido, se muestra un módulo datos regrabables direccionables de recombinasa (RAD), que almacena la información digital de forma fiable dentro de un cromosoma. RAD usa módulos de serina de la integrasa y las funciones de excision se adaptan a partir de bacteriófagos para invertir y recuperar secuencias específicas de ADN.
Nuestro elemento de memoria núcleo RAD es capaz de almacenar información pasiva en ausencia de expresión de genes heterólogos por más de 100 divisiones celulares y se puede conmutar repetidamente sin degradación del rendimiento, como se requiere para soportar el almacenamiento de datos. También demuestran cómo la estocasticidad programada en el rendimiento del sistema RAD derivada de la recombinación bidireccional se puede lograr y ajustar por la variación de las tasas de síntesis y degradación de la proteína recombinasa. Las funciones serina recombinasa utilizadas aquí no requieren células específicas de cofactores y deberían ser útiles en la extensión de los métodos de cómputo y control para el estudio e ingeniería de muchos sistemas biológicos.
Ampliar en: Rewritable digital data storage in live cells via engineered control of recombination directionality
Científicos de Berkeley Lab generan electricidad a partir de virus
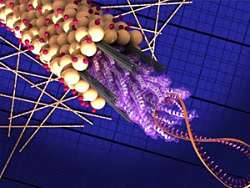
Un nuevo enfoque es un primer paso prometedor hacia el desarrollo de pequeños dispositivos que capten energía eléctrica de las tareas cotidianas. Imagínate cargar el teléfono mientras caminas, gracias a un generador de papel delgado incrustado en la suela del zapato. Este escenario futurista es ahora un poco más real. Científicos del Lawrence Berkeley National Laboratory (Berkeley Lab) del Departamento de Energía de EE.UU. han desarrollado un método para generar energía utilizando virus inofensivos que convierten la energía mecánica en electricidad.
Los científicos probaron su enfoque mediante la creación de un generador que produce la corriente necesaria para operar una pequeña pantalla de cristal líquido. Funciona pulsando con un dedo en un electrodo del tamaño de un sello de correos revestido con virus especialmente diseñados. Los virus convierten la fuerza en una carga eléctrica.
El generador es el primero en producir electricidad mediante el aprovechamiento de las propiedades piezoeléctricas de un material biológico. Piezoelectricidad es la acumulación de una carga en un sólido en respuesta a una tensión mecánica.
El hito podría dar lugar a pequeños dispositivos que recolectan energía eléctrica a partir de las vibraciones de las tareas cotidianas, como el cierre de una puerta o subir escaleras.
También apunta a una forma más sencilla de hacer los dispositivos microelectrónicos. Eso es porque los virus se organizan en una película ordenada que permite que el generador funcione. El autoensamblaje es una gran meta perseguida en el meticuloso mundo de la nanotecnología.
Los científicos describen su trabajo en una publicación adelantada en internet el 13 de mayo, de la revista Nature Nanotechnology.
«Se necesita más investigación, pero nuestro trabajo es un primer paso prometedor hacia el desarrollo de generadores de energía personales, actuadores para su uso en nanodispositivos, y otros dispositivos basados en electrónica de virus», dice Seung-Wuk Lee, un científico de la División de Física del Laboratorio de Biociencias de la Universidad de Berkeley y profesor asociado de bioingeniería.
Él condujo la investigación con un equipo que incluye Ramamoorthy Ramesh, un científico de materiales del laboratorio de Berkeley División de Ciencias y profesor de ciencias de los materiales, la ingeniería y la física en UC Berkeley, y Yang Lee Byung de la División de Física del Laboratorio Berkeley Biociencias.
El efecto piezoeléctrico fue descubierto en 1880 y desde entonces ha sido encontrado en los cristales, cerámica, huesos, proteínas y ADN. También se ha puesto en uso en encendedores de cigarrillos eléctricos y los microscopios de sonda no podría funcionar sin él, para nombrar unas pocas aplicaciones.
Sin embargo, los materiales utilizados para fabricar dispositivos piezoeléctricos son tóxicos y muy difícil de trabajar, lo que limita el uso generalizado de esta tecnología.
Lee y sus colegas se preguntaban si un virus estudiado en los laboratorios de todo el mundo ofrece una mejor forma. El bacteriófago M13 sólo ataca a las bacterias y es benigno para las personas. Al ser un virus, se reproduce por millones en cuestión de horas, así que siempre hay un suministro constante. Es fácil de manipular genéticamente. Y un gran número de los virus en forma de varilla, naturalmente, se orientan bien ordenados en películas, tanto como la forma en que los palillos se alinean en una caja.
Estos son los rasgos que los científicos buscan en un nanobloque de construcción. Sin embargo, los investigadores de Berkeley Lab primero tenía que determinar si el virus M13 es piezoeléctrico. Lee acudió a Ramesh, un experto en el estudio de las propiedades eléctricas de películas delgadas a nanoescala. Aplicó un campo eléctrico a una película de virus M13 y vio lo que pasó en un microscopio especial. Proteínas helicoidales que envuelven los virus se retorcían y giraban en respuesta, una señal segura del efecto piezoeléctrico en el trabajo.
Los científicos precisan una mayor fuerza piezoeléctricos del virus. Se utiliza ingeniería genética para añadir cuatro residuos de aminoácidos cargados negativamente a un extremo de las proteínas helicoidales que recubren el virus.Estos residuos aumentan la diferencia de carga entre los extremos de las proteínas positivas y negativas, lo que aumenta la tensión del virus.
Los científicos han mejorado aún más el sistema por el apilamiento de películas compuestas de capas individuales del virus una encima del otro. Se encontró que una pila de aproximadamente 20 capas de espesor mostraba el efecto piezoeléctrico más fuerte.
Lo único que quedaba por hacer era una prueba de demostración, así que los científicos fabricaron un generador de virus basada en energía piezoeléctrica. Ellos crearon las condiciones para los virus modificados genéticamente para organizar de forma espontánea en una película de capas múltiples que mide aproximadamente un centímetro cuadrado. Esta película se intercaló entonces entre dos electrodos revestidos de oro, que estaban conectados por cables a una pantalla de cristal líquido.
Cuando se aplica presión en el generador, que produce un máximo de seis nanoamperios de corriente y 400 milivoltios de potencial. Eso es suficiente corriente para parpadear el número «1» en la pantalla, y aproximadamente una cuarta parte de la tensión de una batería triple A .
«Ahora estamos trabajando en maneras de mejorar en esta demostración de la prueba de principio», dice Lee. «Debido a que las herramientas de la biotecnología permitirán a gran escala la producción de virus modificados genéticamente, los materiales piezoeléctricos sobre la base de los virus podrían ofrecer una ruta sencilla a una nueva microelectrónica en el futuro.»
Fuente: EurekAlert!
Las abejas son capaces de manipular ideas abstractas

Un estudio realizado por investigadores del CNRS (Francia) pone de relieve la capacidad del cerebro de las abejas para considerar las relaciones entre los objetos, como su lugar en algunas otras funciones.
Las abejas, como los mamíferos, son capaces de ver los objetos entre sí sobre la base de tomar en cuenta las relaciones espaciales, tales como: «por encima», «abajo» o «siguiente». Un estudio realizado por un equipo de investigadores del CNRS dirigido por Martin Giurfa, constituye la sorprendente capacidad de estos insectos para generar y manipular los conceptos con el fin de obtener acceso a los recursos nutricionales.
Al llegar a estas conclusiones, los investigadores realizaron una serie de experimentos en los que las abejas ofrecen dos agujeros que conducen a un tanque de agua similar a una dulce recompensa o un líquido similar a un amargo castigo. El orificio que conduce a la recompensa fue, por otra parte, indicada por una serie de imágenes diferentes, pero siempre en la misma relación (por ejemplo, por debajo del agujero).
Los resultados publicados en la revista Proceedings de la Academia Nacional de Ciencias indican que después de una treintena de ensayos, las abejas son capaces de reconocer una relación inmediata que indica que el agua dulce, a pesar de que las imágenes difieren pruebas. «Lo que es notable es que se pueden utilizar dos diferentes conceptos para tomar una decisión ante una nueva situación», dice Martin Giurfa en comentarios informados por Le Parisien.
Los investigadores dijeron que el nuevo estudio destruye por completo muchas teorías en campos tan diversos como la cognición animal, la psicología humana, las neurociencias o la inteligencia artificial.
Fuente: MaxiSciences
La teoría matemática de la comunicación de Shannon aplicada al secuenciado de ADN
Nadie sabe qué tecnología de secuenciado es más rápida debido a que nunca ha habido una forma justa de comparar las tasas a las que se extrae información del ADN. Hasta ahora.
Uno de los grandes héroes desconocidos de la ciencia del siglo XX es Claude Shannon, ingeniero de los famosos Laboratorios Bell durante su auge en la mitad del siglo XX. La más perdurable contribución a la ciencia por parte de Shannon es su teoría de la información: la idea que apuntala toda la comunicación digital.
En un famoso artículo que data de finales de la década de 1940, Shannon fijó el problema fundamental de la comunicación: reproducir en un punto del espacio un mensaje que se había creado en otro punto. El mensaje se codificaba inicialmente de alguna manera, se transmitía, y luego se decodificaba.
Shannon demostró que un mensaje siempre puede reproducirse en otro punto del espacio con una precisión arbitraria siempre que el ruido esté por debajo de un nivel umbral. Pasó luego a calcular cuánta información podría enviarse de esta forma, una propiedad conocida como capacidad del canal de información.
Las ideas de Shannon se han aplicado ampliamente a todas las formas de transmisión de información con gran éxito. Una vía particularmente interesante ha sido la aplicación de la teoría de la información a la biología – la idea de que la propia vida es la transmisión de información de una generación a la siguiente.
Este tipo de pensamiento revolucionario está en proceso y aún en sus primeras etapas. Queda mucho por llegar.
Hoy revisamos un interesante corolario en el área de la transmisión de información biológica. Abolfazl Motahari y sus colegas de la Universidad de California en Berkeley, usan la aproximación de Shannon para examinar cómo de rápidamente puede extraerse la información del ADN usando el proceso del secuenciado de escopeta.
El problema aquí es determinar la secuencia de nucleótidos (A,G,C y T) en un genoma. Esto requiere tiempo debido a que los genomas tienden a ser largos – por ejemplo, el genoma humano consta de unos 3000 millones de nucleótidos o pares de bases. Secuenciar esta cantidad en serie llevaría una infinidad de tiempo.
La aproximación de escopeta implica cortar el genoma en trozos aleatorios, que constan de entre 100 y 1000 bases, y secuenciarlas en paralelo. La información se vuelve a pegar in silico mediante un algoritmo conocido como de re-ensamblado.
Por supuesto, no hay forma de saber cómo re-ensamblar la información procedente de una única ‘lectura’ del genoma. Por lo que en la aproximación de escopeta, este proceso se repite muchas veces. Dado que cada lectura divide el genoma de una forma distinta, los otros inevitablemente se solapan con segmentos de la ejecución anterior. Estas áreas de solapamiento hacen posible el re-ensamblado de todo el genoma, como un rompecabezas.
Esto tiene el aspecto del problema clásico de la teoría de la información y, efectivamente, distintas personas han pensado en ello de esta forma. Sin embargo, Motahari y compañía van un paso más allá cambiando su enunciado más o menos exactamente a un análogo de la famosa aproximación de Shannon.
Dicen que el problema del secuenciado del genoma es esencialmente la reproducción de un mensaje escrito en el ADN a un formato electrónico digital. Según esta forma de abordarlo, el mensaje original está en el ADN, se codifica para su transmisión mediante el proceso de lectura y luego se decodifica por el algoritmo de re-ensamblado para producir una versión electrónica.
Lo que demuestran es que hay una capacidad del canal que define una tasa máxima para el flujo de información durante el proceso de secuenciado. “Ofrece el número máximo de pares de bases de ADN que pueden resolverse en cada lectura, mediante cualquier algoritmo de ensamblaje, sin importar las limitaciones computacionales”, comentan.
Esto es un resultado significativo para cualquiera interesado en el secuenciado de genomas. Un tema importante es lo rápido que una tecnología concreta de secuenciado puede realizar esta tarea, y si es más rápida o lenta que otras aproximaciones.
Por el momento no es posible calcularlo debido a que muchos de los algoritmos usados para ensamblado están diseñados para tecnologías y aproximaciones específicas a la lectura. Motohari y sus colegas dicen que hay, al menos, 20 algoritmos distintos de re-ensamblado, por ejemplo. “Esto hace difícil comparar distintos algoritmos”, comentan.
Por consiguiente, nadie sabe realmente cuál es más rápido, o incluso cuál tiene el potencial de ser más rápido.
El nuevo trabajo cambia esto. Por primera vez debería ser posible calcular lo cerca que está una tecnología concreta de secuenciado del límite teórico.
Esto podría forzar una limpieza de la madera muerta en esta área y estimular un periodo de innovación rápida en la tecnología del secuenciado.
Artículo de Referencia: arxiv.org/abs/1203.6233: Information Theory of DNA Sequencing
Traducido en: Ciencia Kanija
Transistores biodegradables
 Ganadora de un premio de investigación, la Universidad de Tel Aviv (Israel) utiliza automontaje de sangre, leche y proteínas del moco para construir la próxima generación de tecnología
Ganadora de un premio de investigación, la Universidad de Tel Aviv (Israel) utiliza automontaje de sangre, leche y proteínas del moco para construir la próxima generación de tecnología
El silicio, un elemento semiconductor, es la base de la más moderna tecnología, incluyendo teléfonos móviles celulares y ordenadores. Sin embargo, según investigadores de la Universidad de Tel Aviv, este material se está convirtiendo rápidamente en una industria obsoleta, pues la tendencia es hacia la producción de componentes cada vez más pequeños y que sean menos perjudiciales para el medio ambiente.
Un equipo que incluye a los estudiantes de doctorado Elad Mentovich y Hendler Netta del Departamento de Química de la UTA y el Centro de Nanociencia y Nanotecnología, con el supervisor el Dr. Richter Shachar y en colaboración con el Prof. Michael Gozin y su estudiante de doctorado Bogdan Belgorodsky, ha reunido las técnicas más avanzadas de múltiples campos de la ciencia para crear transistores basados en proteínas, semiconductores utilizados para alimentar dispositivos electrónicos, desde los materiales orgánicos que se encuentran en el cuerpo humano. Podrían convertirse en la base de una nueva generación del tamaño de las nanotecnologías, que sean flexibles y biodegradables.
Trabajan con las proteínas de la sangre, leche, y el moco, que tienen la capacidad de autoensamblarse para formar una película semiconductora, los investigadores ya han logrado dar el primer paso hacia las pantallas biodegradables, y su objetivo es utilizar este método para desarrollar todos los dispositivos de la electrónica. Su investigación, que ha aparecido en la revista Nano Letters and Advanced Materials, recibió recientemente la medalla de plata en los Materials Research Society Graduate Student Awards en Boston, MA.
Construyendo el mejor transistor de abajo hacia arriba
Uno de los retos de la utilización de silicio como semiconductor es que un transistor se debe crear mediante un «arriba hacia abajo». El fabricante ha de empezar con una oblea de silicio y esculpir la forma que se necesita, como tallar una escultura a partir de una roca. Este método limita las capacidades de los transistores cuando se trata de factores tales como el tamaño y flexibilidad.
Los investigadores se volvieron hacia la biología y la química, con un enfoque diferente para la construcción del transistor ideal. Cuando apilaban varias combinaciones de sangre, leche y proteínas mucosas a cualquier material de base, las moléculas se autoensamblaban para crear una película semiconductora a nanoescala. En el caso de proteína de la sangre, por ejemplo, la película es aproximadamente de cuatro nanómetros alta. La tecnología actual en uso ahora es de 18 nanómetros, dice Mentovich.
Juntos, los tres tipos diferentes de proteínas crenr un circuito completo con capacidades electrónicas y ópticas, cada uno trayendo algo único al dispositivo. La proteína de la sangre tiene la capacidad para absorber oxígeno, Mentovich dice, que permite el «dopaje» de semiconductores con productos químicos específicos a fin de crear propiedades tecnológicas específicas. Las proteínas de la leche, conocidas por su fortaleza en entornos difíciles, forman las fibras, que son los componentes básicos de los transistores, mientras que las proteínas de la mucosa tiene la capacidad de mantener de color rojo, verde y azul por separado tintes fluorescentes, así como la creación de la emisión de luz blanca que es necesaria para la óptica avanzada.
En general, las habilidades naturales de cada proteína dan a los investigadores «control único» sobre el transistor orgánico resultante, lo que permite ajustes para la conductividad, el almacenamiento de memoria, y la fluorescencia entre otras características.
Una nueva era de la tecnología
La tecnología está ahora cambiando de una época de silicio a una época de carbono, según Mentovich, y este nuevo tipo de transistor podría desempeñar un papel importante. Los transistores construidos a partir de estas proteínas son ideales para los circuitos más pequeños y flexibles, que están hechos de plástico en lugar de silicio, que existe en forma de oblea que se rompen como el cristal si se doblan. El descubrimiento podría conducir a una nueva gama de tecnologías flexibles, como pantallas, teléfonos celulares, tabletas, biosensores, y hips de microprocesadores.
Tan significativo es, que debido a que los investigadores están utilizando proteínas naturales para construir su transistor, los productos que crean serán biodegradables. Es una tecnología mucho más respetuosa con el medio ambiente que aborda el creciente problema de la basura electrónica, que desborda los vertederos de todo el mundo.
Fuente: EurekAlert!
¿Las pantallas del futuro van a estar compuestas de bacterias?
Investigadores acaban de completar la hazaña de hacer un panel de luz intermitente integrado por … bacterias .
En un momento en todo el mundo habla de píxeles para los televisores, ordenadores y modernas cámaras, es el momento de introducir el concepto de «biopixels«.
La revista Nature publicó un artículo con el trabajo de un increíble equipo de investigadores compuesto por Arthur Prindle, Samayoa Phillip, Razinkov Ivan, Dañino Tal, Lev S. Tsimring y Jeff apresurada. Todos ellos pertenecen a distintos laboratorios de la Universidad de California en San Diego (EE.UU.). Este equipo ha conseguido que paneles de luz formado por millones de bacterias sean capaces de brillar de manera constante y al mismo tiempo.
Para lograr sus fines, las proteínas fluorescentes se adjuntaron a «relojes biológicos» de cada una de las bacterias del aparato. Estos se dividieron en varias colonias. El siguiente paso fue para sincronizar todos los relojes, lo que permite todas las células vivas de la misma unidad que produce la luz al mismo tiempo. Finalmente, todas las colonias fueron sincronizados entre sí. El resultado final ofrece miles de juegos de bacterias, se compone de cientos de unidades de vida, capaces de brillar al mismo tiempo. Sólo queda que estas unidades de luz se ubiquen sobre una placa y se obtiene el panel deseado.
Este video (en Inglés) presenta las diferentes etapas de desarrollo de paneles de luz formado por millones de bacterias. También se pueden ver ejemplos de las operaciones y algunas aplicaciones prácticas. © UCsandiego / YouTube.
Paneles bacterianos intermitentes ¿Para qué?
Más allá de los logros técnicos representados por este descubrimiento, los autores destacaron la utilidad que su dispositivo puede tener en la vida ordinaria. Los paneles pueden ser utilizados como biosensores. Proporcionan información sobre la presencia o ausencia de diversos contaminantes, sustancias tóxicas o microorganismos patógenos. Que dependerá de la sensibilidad de las bacterias frente a estos agentes. El alcance y el ritmo de disminución del parpadeo, después, informará sobre el nivel de toxicidad de los elementos detectados en tiempo real. Como prueba, se ha desarrollado un dispositivo para la detección de contaminación del aire por arsénico. Los sensores muestran otra de las ventajas biológicas: que se ejecutan continuamente en el momento en que muchos sistemas de detección de químicos pueden ser utilizados sólo ocasionalmente.
Ampliar información en: Futura Sciences
Vida eterna, transfiriendo nuestro cerebro a un ordenador
Ray Kurzweil y otros llamados transhumanistas han prometido que en las próximas décadas vamos a ser capaces de transferir una copia digital de los trillones de conexiones entre las células nerviosas del cerebro en un ordenador. En esencia se nos reencarnará como seres no biológicos, que persisten durante toda la eternidad dentro de un ordenador portátil, en los enlaces de internet o como avatares en el interior de un televisor. Después de lograr la copia final y pegar, sería decir adiós a la muerte tal como la conocemos.
Por razones bastante evidentes, los biólogos tienden a rechazar de plano las ideas de Kurzweil y muchos transhumanistas, como si éstas fueran los desvaríos de deportistas en equipo que no saben nada acerca del funcionamiento real del ADN y las células que componen los tejidos vivos. En este debate aparee Sebastian Seung, un neurocientífico computacional joven y bien considerado en el MIT, que ha tomado con seriedad algunas de las preguntas planteadas por los transhumanistas.
En Conectome, Seung transmite la emoción de estudiar el esquema completo del cerebro que da nombre al libro. Un conectoma completo podría proporcionar idea de lo que va mal en el cerebro, por ejemplo, en un niño autista o en un paciente de Alzheimer (definitivamente vale la pena leer estos bits solamente). En los últimos capítulos, a pesar de que se ocupa las ideas de los transhumanistas que desesperadamente quieren tener en sus manos un conectoma completo para la subida binaria a la inmortalidad final.
Seung trata de luchar a brazo partido con la afirmación controvertida de que algún día podría ser capaz de transferir el equivalente de un archivo connectome.doc de hardware, software a cualquier otro robot o avatar que usted pueda escoger.
Seung muestra una pose que mezcla escepticismo con fascinación. La copia de la lectura del avance que la revista Scientific American ha recibido muestra algunas dudas:
«En su libro Vivir lo suficiente para vivir para siempre, el inventor Ray Kurzweil predice que la inmortalidad se alcanzará en las próximas décadas», dice Seung. «Si usted puede llegar a vivir lo suficiente para sobrevivir a ese punto, va a vivir para siempre. Personalmente, me siento bastante seguro de que ustedes, queridos lectores, van a morir, y yo también »
Pero sigue estando Seung intrigado por la idea de que un mecanismo de unificación de las unidades de funcionamiento de la máquina de carne entre nuestros oídos y su mecánica puede ser descifrable y reproducible. Y está por lo menos dispuesto a dirigir una mirada crítica sobre la posibilidad de una versión 2.0 de sí mismo que, cuando se transfiera a un superordenador, ordenador portátil, o avatar de software, entonces puede vivir como un fantasma electrónico. (Sí, algunos dirían que Facebook ya nos ha llevado hasta allí, pero Seung no se ocupa de los medios de comunicación social, como la inmortalidad.)
La pregunta central de Seung -y que también mantiene en vilo a los transhumanistas- es si usted es su conectoma. ¿Si usted pudiera deducir todos los puntos de conexión de todas las células del cerebro, la fuerza con la que cada neurona se activa, y la forma en que estos patrones de activación cambian a medida que las células interactúan entre sí, que de hecho, que se quedará con una copia de usted?
En un capítulo titulado «Para congelar o Pickle» Seung se compromete, desde múltiples perspectivas, con un análisis serio del conectoma como un camino hacia la inmortalidad. Todas sus conclusiones apuntan a los obstáculos que podrían muy bien resultar insuperables.
En primer lugar se considera lo que llama el problema de meatlocker. Debido a que puede tardar un tiempo crear el diagrama completo del cableado, muchos transhumanistas tienen planes de colocar sus cabezas o cuerpos enteros en un recipiente Dewar criogénico de nitrógeno líquido pronto después de la muert e-o, como alternativa, para conservarse en un sólido cristalino o por otro proceso llamado plastinación. (La plastinación es la forma de conservación utilizados en la gira de Body Worlds de cadáveres sin piel).
Una vez que las tecnologías se perfeccionan, la idea vaa más, el tejido preservado podría ser utilizado para recomponer el esquema de conexión. Por sí sola, esta expectativa puede ser un atolladero debido a la dificultad de mantener la integridad del circuito de insondable complejidad del cerebro. «En la actualidad, la criónica se acerca más a la religión que a la ciencia», Seung escribe. «Sus miembros creen que una civilización futura será capaz de resucitarlos, basándose únicamente en su fe en el progreso tecnológico sin límites.»
Incluso si este detalle insignificantes pueden llegar a ser resuelto, queda la asignatura pendiente de la información que el conectoma contiene exactamente. Para entender mejor las conexiones del cerebro, los científicos han estado tratando de simular las partes del cerebro durante décadas. Ahora están tomando también la cuestión más amplia de recrearlo todo. El Proyecto Cerebro Humano en Europa se ha centrado en la tarea de elaborar un modelo de todo el órgano en una década. El modelo, en principio, consiste en simular los miles de tipos de neuronas diferentes, así como las conexiones entre ellas y sus estructuras cambiando a medida que el cerebro aprende y se olvida.
El Proyecto Cerebro Humano pretende ser una exploración de la ciencia básica, no una preparación para la vida eterna. Sin embargo, apunta Seung que incluso un esfuerzo impresionante de su magnitud podría dejar de captar toda la información necesaria.
Una falla potencial: El modelo del cerebro podría tener que tomar en cuenta la forma en que las neuronas se comunican fuera de los canales conocidos de la transmisión de señales químicas y eléctricas a través de pequeños espacios, llamadas sinapsis, entre las células cerebrales. Para superar esta dificultad, puede ser necesario crear una simulación de cada átomo en el cerebro, una empresa de una complejidad inimaginable tal que sería rayar en lo imposible. «Me parece absurdo siquiera considerar el enorme poder de cómputo necesario, y está completamente fuera de la cuestión a menos que su descendencia sobreviva a distancia para escalas de tiempo galácticas», escribe.
Seung termina su libro con un epílogo que aboga por una «vuelta a la realidad», un reconocimiento de que los «grandes retos» siguen siendo, más allá de las misiones quijotesca para la vida eterna. Un esfuerzo de 10 años para encontrar el conectoma de un cerebro de ratón se encuentra en su lista de deseos. Tal búsqueda no tiene el atractivo de la contemplación de la eternidad como un archivo en una unidad flash. En el final, sin embargo, Seung cree en un proyecto de esta escala más modesta, al igual que el Proyecto Genoma Humano que los investigadores empujan hasta el límite, pero se ha de profundizar mucho en el conocimiento acerca de un órgano que sigue siendo un misterio.
Fuente: Aleksandar Zlateski y Sebastian Seung
¿Un ordenador cuántico en el ojo del pájaro petirrojo?

“El petirrojo es un ave migratoria que utiliza como brújula magnética unas proteínas llamadas criptocromos situadas en la retina de sus ojos, según el biólogo Wolfgang Wiltschko (Universidad de Frankfurt). El físico cuántico Vlatko Vedral (Universidad de Oxford) cree que en el criptocromo se entrelazan cuánticamente varios electrones cuyos espines actúan como cubits. Según este especialista en computación cuántica, la Naturaleza ha logrado fabricar el primer computador cuántico, mucho antes que los propios físicos. La Naturaleza va por delante de los investigadores más osados y quizás el primer computador cuántico sea una versión biomimética del criptocromo. (…) La fuente de la noticia es un artículo de Vedral publicado en Physical Review Letters (PRL). Un artículo en PRL es, a priori, una gran garantía de calidad científica. ¡Increíble! Efectos cuánticos en la brújula magnética de las aves migratorias y en particular en el petirrojo (Erithacus rubecula). (…) Increíble, pero yo no me lo creo. Como físico no me puedo creer que el entrelazamiento cuántico de los espines de pares de electrones se mantenga en una proteína a temperatura ambiente durante 20 microsegundos. En mi opinión, es un tiempo millones de veces más largo de lo razonable a temperatura ambiente.
Ampliar en: Amazing.es
Puertas lógicas que hacen más fácil la computación con ADN
Los ordenadores fabricados a partir del ADN se beneficiarán de un gran avance en el diseño, que les ayudará a realizar cálculos complejos, y también podría dar lugar a sensores biológicos para detectar enfermedades.
Hasta ahora, los ordenadores de ADN – que utilizan cadenas de moléculas de ADN para almacenar datos y procesarlos – se construyen básicamente de forma manual, sus diseñadores eligen las estructuras de ADN exactas necesarias para los circuitos lógicos precisos para el cálculo. Ahora, Lulu Qian y Winfree Erik en el Instituto de Tecnología de California en Pasadena (EE.UU.) han desarrollado nuevas puertas lógicas «see-saw» que permiten automatizar el proceso, por lo que es posible construir circuitos mucho más grandes.
Los investigadores utilizaron estas puertas para construir un circuito que calcula la raíz cuadrada de 0, 1, 4 y 9. Se compone de 130 cadenas de ADN, la mayor construida en un tubo de ensayo. «Esto en sí mismo no es el logro – sino que es una especie de confirmación de los principios en que nuestros diseños se basan», indica Winfree. «Si se puede conseguir un circuito para hacer algo tan arbitrario y ajeno a la química como calcular la raíz cuadrada, a continuación, probablemente se podrán conseguir circuitos de ADN para hacer cualquier cosa.»
Fuera de la edad del código máquina
Su enfoque imita el desarrollo de los ordenadores convencionales basados en el silicio, que fueron programadas originalmente en código máquina, un conjunto de bajo nivel de instrucciones tan detalladas que es casi imposible de leer para los seres humanos. Los ingenieros de software utilizan modernos lenguajes de programación de alto nivel de y la ejecución a través de un software llamado compilador, que convierte los comandos en instrucciones de nivel adecuado a la electrónica de la computadora. Los programadores de ADN será ahora capaz de hacer lo mismo, con el diseño de sus circuitos en lógica de alto nivel sin tener que preocuparse acerca de las moléculas subyacentes. «Básicamente tenemos un compilador rudimentaria para los circuitos moleculares», dice Winfree.
Qian y Winfree afirman que sus puertas «see-saw» (sube y baja) fueron la clave para el desarrollo de su compilador. Actúan como interruptores, convirtiendo una señal de ADN a otra; un par puede realizar las operaciones lógicas Y (and) u O (or). La combinación de estas operaciones de una manera particular, conocida como la lógica de «dual rail» (rail dual), permite calcular efectivamente cualquier cosa con un circuito de ADN.
Hay algunas limitaciones, las señales no pueden viajar hacia atrás a lo largo del circuito, por lo que es imposible crear una memoria para almacenar valores. Winfree dice que debido a esta y otras limitaciones, muachos circuitos ADN seguirán por ahor siendo construidos «a mano».
«En cierto sentido, están abriendo la puerta a la materia programable ![]() «, Dice Martyn Amos , un experto en computación con ADN en el Manchester Metropolitan University (Reino Unido). Afirma que los ordenadores de ADN basados en el nuevo método es poco probable que sean rivales frente a sus homólogos de silicio, pero podrían ser mucho mejores en el análisis de materiales biológicos. «Usted puede imaginarse el introducir una muestra en un tubo, agitar y dejarlo reposar por unas horas. Eso es preferible que el envío a un laboratorio y pegarla en una máquina. »
«, Dice Martyn Amos , un experto en computación con ADN en el Manchester Metropolitan University (Reino Unido). Afirma que los ordenadores de ADN basados en el nuevo método es poco probable que sean rivales frente a sus homólogos de silicio, pero podrían ser mucho mejores en el análisis de materiales biológicos. «Usted puede imaginarse el introducir una muestra en un tubo, agitar y dejarlo reposar por unas horas. Eso es preferible que el envío a un laboratorio y pegarla en una máquina. »
Fuente: Sience , DOI: 10.1126/science.1200520

autobus las palmas aeropuerto cetona de frambuesa